

 2024-09-05
2024-09-05 776
776
2024年9月4日23时(北京时间),复旦大学公共卫生学院、上海市重大传染病和生物安全研究院粟硕团队与合作者在《自然》(Nature)杂志发表题为“Farmed fur animals harbor viruses with zoonotic spillover potential”的研究性论文,应用前沿交叉研究方法揭示多种养殖哺乳动物中的高分辨率病毒基因组序列组成、潜在跨物种传播风险和生态学特征,将为构建多维度公共卫生风险评估与新发传染病预测预报体系提供数据支持。《自然》杂志同日配发研究亮点(Highlight),给予该研究高度评价。

野生与养殖哺乳动物携带高度多样性的病原微生物,是新发病原体进一步传播的重要枢纽。近期国内外多项研究表明,鼩鼱等啮齿目及水貂等养殖食肉目动物具有携带亨尼帕病毒、冠状病毒和禽流感病毒等人畜共患病毒的潜力,对动物源病毒的探索是“同一个健康”框架下预测预警新发传染病的基础(Zhang JT et al. (2024), Agüero et al. (2022), Lu L et al. (2021))。
当前哺乳动物种类繁多,除猪等传统家畜外,缺乏对其他动物尤其是养殖哺乳动物多地域、多类群、跨物种等不同维度内的综合病毒组挖掘与跨尺度信息整合研究。因此,这些动物的病原生态学与流行病学数据存在较大空白,并成为新发传染病疫情精准预测预报与公共卫生高效监测管理的重要瓶颈。
粟硕团队与合作者对来自食肉目、偶蹄目等多种可用于皮毛、药物等用途的哺乳动物进行了系统的病毒组研究。团队采用前沿研究方法高分辨率表征了125种脊椎动物相关病毒基因组序列,极大丰富了病毒的多样性和已知病毒的宿主谱。基于单样本建库策略,团队解析了多种病毒在宿主个体水平上的混合感染情况,并进一步通过结合生态学地理分布、进化动力学及公共大数据等多学科交叉研究方法,推定出诺如病毒、盖塔病毒等多种频繁发生“宿主跳跃”的潜在“风险”病毒。同时,团队还从空间聚类,动物类群、种群、组织等多维度揭示了潜在“风险”病毒的生态学与流行病学特征。
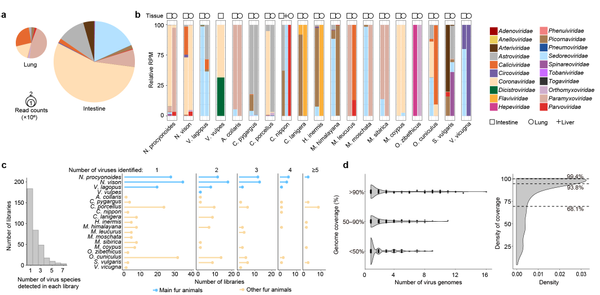
该成果将极大丰富动物源病毒遗传本底数据,为人-动物-环境一体化下建立多维度的新发传染病风险评估体系与新发传染病预测预报奠定重要数据基础。
联系我们
地址:上海市医学院路138号
邮编:200032
Email:yxky@fudan.edu.cn

复旦上医科技

复旦上医
Copyright © 2024 复旦大学上海医学院科研处版权所有

